R语言进阶绘图:生存曲线
两组间带置信区间的生存曲线
以下是一个使用R语言和survival及ggplot2包绘制两组间带置信区间的生存曲线的示例。我们将使用lung数据集,该数据集包含了一组肺癌患者的生存时间和其他相关信息。在此示例中,我们将根据患者是否接受了放疗(变量名为ph.ecog)将数据分为两组。
首先,安装并加载所需的包:
if (!requireNamespace("survival", quietly = TRUE))
install.packages("survival")
library(survival)if (!requireNamespace("ggplot2", quietly = TRUE))
install.packages("ggplot2")
library(ggplot2)if (!requireNamespace("survminer", quietly = TRUE))
install.packages("survminer")
library(survminer)
接下来,加载数据并对数据进行预处理:
# 加载数据
data("lung")
我们展示lung数据集的前几行:
# 显示数据集的前6行
head(lung)
# 将ph.ecog变量转换为因子类型,使其更易于处理 lung$ph.ecog <- as.factor(lung$ph.ecog)
#现在,使用survfit()函数拟合生存曲线模型:
# 使用Cox比例风险模型拟合生存曲线
fit <- survfit(Surv(time, status) ~ ph.ecog, data = lung)
#最后,使用ggsurvplot()函数绘制生存曲线图:
# 绘制生存曲线
p <- ggsurvplot(fit, data = lung,
risk.table = TRUE, # 在图下方显示风险表
pval = TRUE, # 显示log-rank检验的p值
conf.int = TRUE, # 添加置信区间
xlab = "时间(天)", # x轴标签
ylab = "生存概率", # y轴标签
title = "两组患者的生存曲线", # 图标题
legend.labs = c("ECOG 0", "ECOG 1", "ECOG 2", "ECOG 3"), # 图例标签
legend.title = "治疗情况", # 图例标题
linetype = c("solid", "dashed", "dotted", "dotdash"), # 线型
palette = c("#E69F00", "#56B4E9", "#009E73", "#F0E442"), # 颜色
ggtheme = theme_minimal()) # 图形主题# 显示图形
print(p)
在这个示例中,我们首先加载了所需的包和数据,然后对数据进行了预处理。接下来,我们使用了Cox比例风险模型拟合生存曲线,并使用ggsurvplot()函数绘制了生存曲线图。我们设置了各种参数,使图形尽可能美观和详尽,包括添加风险表、显示log-rank检验的p值、添加置信区间以及设置轴标签、图例和线型等。

接下来,我们按照之前的步骤创建生存曲线图。然后,我们将图形保存为PDF文件:
# 保存图形为PDF文件
ggsave(filename = "survival_curve.pdf", plot = p$plot, device = "pdf", width = 10, height = 7, units = "in")
这段代码将在当前工作目录下创建一个名为survival_curve.pdf的文件,其中包含生存曲线图。在ggsave()函数中,你可以自定义PDF文件的大小和分辨率,例如设置width、height和units参数。
具有cut-off的生存曲线
首先,我们将使用survival和survminer包创建生存曲线。这里,我将使用内置的lung数据集作为示例。lung数据集是关于肺癌患者的生存数据。
# 安装并加载所需的包
if (!requireNamespace("survival", quietly = TRUE))
install.packages("survival")
if (!requireNamespace("survminer", quietly = TRUE))
install.packages("survminer")library(survival)
library(survminer)# 加载lung数据集
data(lung)# 显示前几行数据
head(lung)# 创建生存对象
surv_object <- Surv(lung$time, lung$status)
# 使用Cox比例风险模型拟合生存数据
# 以性别(sex)作为分组变量
fit <- survfit(surv_object ~ sex, data = lung)
# 绘制生存曲线
g <- ggsurvplot(fit, data = lung,
risk.table = TRUE,
pval = TRUE,
conf.int = TRUE,
legend.labs = c("Male", "Female"),
xlab = "Time (days)",
ylab = "Survival probability",
title = "Survival curves with 95% confidence intervals",
break.time.by = 500,
ggtheme = theme_minimal())
# 在生存曲线上添加cutoff
cutoff <- 1000
g$table <- g$table + geom_vline(xintercept = cutoff, linetype = "dashed", color = "blue")
g$plot <- g$plot + geom_vline(xintercept = cutoff, linetype = "dashed", color = "blue")
# 显示图形
print(g)
# 保存为PDF文件
ggsave("survival_curves.pdf", g$plot, width = 10, height = 6)
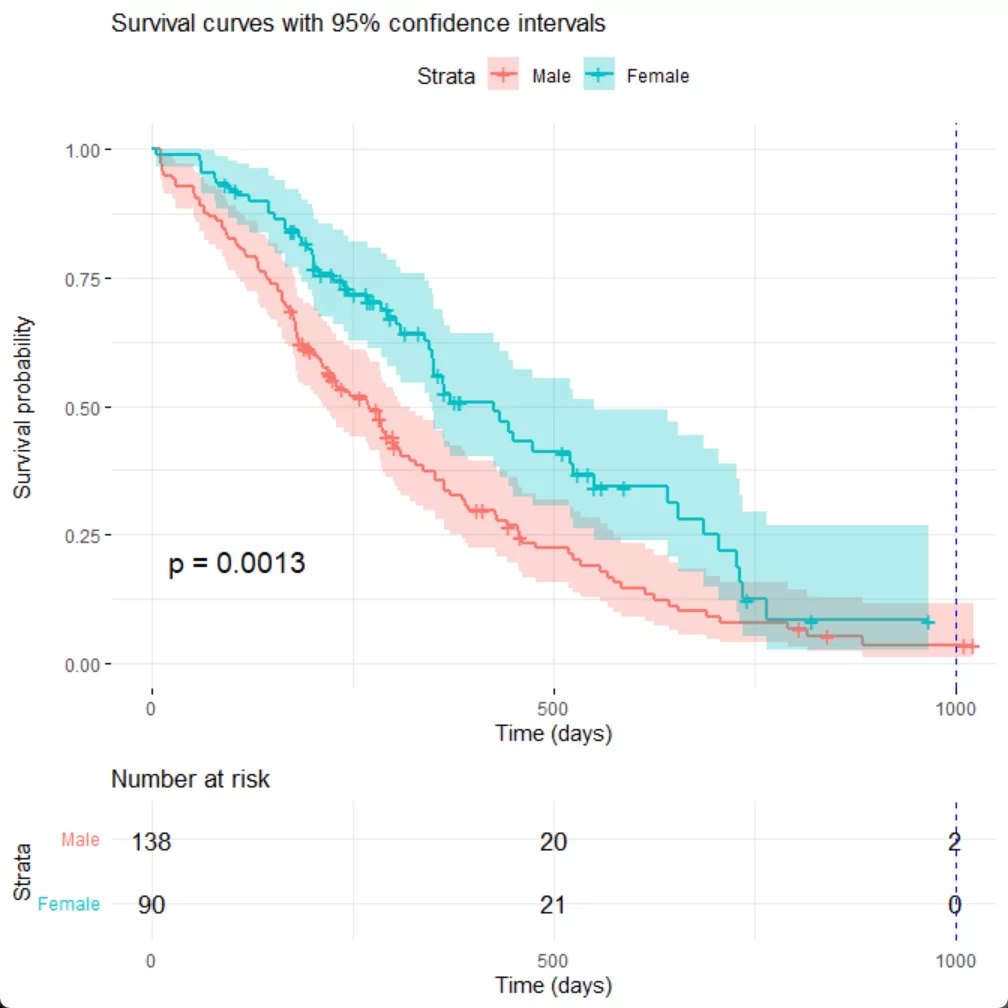
以下是代码解释:
- 加载
survival和survminer包。 - 加载
lung数据集并显示前几行数据。 - 使用
Surv()函数创建生存对象,包含时间和状态。 - 使用
survfit()函数拟合生存数据,以性别(sex)作为分组变量。 - 使用
ggsurvplot()函数绘制生存曲线,添加风险表、置信区间、P值、图例等元素。 - 添加cutoff线,此处使用1000天作为cutoff。
- 显示并保存图形为PDF文件。
两变量组合分组的生存曲线图
为了演示这个例子,我们将使用R中的survival和ggplot2包,并使用lung数据集,这是一个关于肺癌患者生存时间的数据集。lung数据集包含了患者的生存时间(time)、生存状态(status)、年龄(age)和性别(sex)等信息。我们将绘制年龄和性别分组的生存曲线,并添加置信区间和cutoff。
# 安装并加载所需的包
if (!requireNamespace("survival", quietly = TRUE))
install.packages("survival")
library(survival)if (!requireNamespace("ggplot2", quietly = TRUE))
install.packages("ggplot2")
library(ggplot2)if (!requireNamespace("survminer", quietly = TRUE))
install.packages("survminer")
library(survminer)
# 加载lung数据集,并显示前几行
data(lung)
head(lung)
# 创建分组变量 lung$group <- factor( (lung$sex == 1 & lung$age >= 65) * 1 + (lung$sex == 1 & lung$age < 65) * 2 + (lung$sex == 2 & lung$age >= 65) * 3 + (lung$sex == 2 & lung$age < 65) * 4, labels = c("女性 - 高龄", "女性 - 低龄", "男性 - 高龄", "男性 - 低龄") )
# 生存分析
# 使用time(生存时间)和status(生存状态)创建Surv对象
# status = 1表示生存,status = 2表示死亡
lung_surv <- Surv(time = lung$time, event = lung$status == 2)
# 使用Cox比例风险模型进行生存分析
cox_model <- coxph(lung_surv ~ age + sex, data = lung)
# 绘制生存曲线
ggsurvplot(
cox_model, # Cox模型
data = lung, # 数据集
risk.table = TRUE, # 显示风险表
pval = TRUE, # 显示p值
conf.int = TRUE, # 显示置信区间
break.time.by = 100, # 时间刻度间隔
ggtheme = theme_bw(), # 使用黑白主题
xlab = "时间(天)", # x轴标签
ylab = "生存概率", # y轴标签
title = "生存曲线(年龄和性别分组)", # 图标题
legend.title = "组别", # 图例标题
legend.labs = c("女性 - 高龄", "女性 - 低龄", "男性 - 高龄", "男性 - 低龄"), # 图例标签
ylim = c(0, 1), # y轴范围
palette = c("#E69F00", "#56B4E9", "#009E73", "#F0E442"), # 颜色设置
censor = TRUE, # 显示censor点
fontsize = 3, # 字体大小
cutoff = c("age=65", "sex=1") # cutoff设置
) -> plot
# 保存为PDF文件
ggsave(file = "Survival_curve.pdf", plot = plot, width = 10, height = 7)
这个示例将生成一个生存曲线图,展示了年龄和性别分组的生存概率。曲线周围的阴影区域表示置信区间。cutoff值(在这个例子中是年龄=65和性别=1)将数据分为四组:女性-高龄、女性-低龄、男性-高龄和男性-低龄。下面详细解释每一行代码:
- 安装并加载所需的包:
survival、ggplot2和survminer。 - 加载
lung数据集,并显示前几行。 - 使用
lung数据集中的time(生存时间)和status(生存状态)列创建一个Surv对象。 - 使用Cox比例风险模型进行生存分析。模型基于
lung_surv、年龄(age)和性别(sex)。 - 使用
ggsurvplot()函数绘制生存曲线:cox_model:输入的Cox模型。data:数据集。risk.table:是否显示风险表。pval:是否显示p值。conf.int:是否显示置信区间。break.time.by:时间刻度间隔。ggtheme:绘图主题。xlab、ylab:x轴和y轴标签。title:图标题。legend.title:图例标题。legend.labs:图例标签。ylim:y轴范围。palette:曲线颜色设置。censor:是否显示censor点。fontsize:字体大小。cutoff:设置cutoff值(年龄和性别)。
- 使用
ggsave()函数将图保存为PDF文件,指定文件名、绘图对象和图片尺寸。
这个示例展示了如何使用R语言绘制生存曲线,并根据年龄和性别分组。图中还显示了置信区间和cutoff值。