基因相关性圈图(Circos图)
1.图片特点
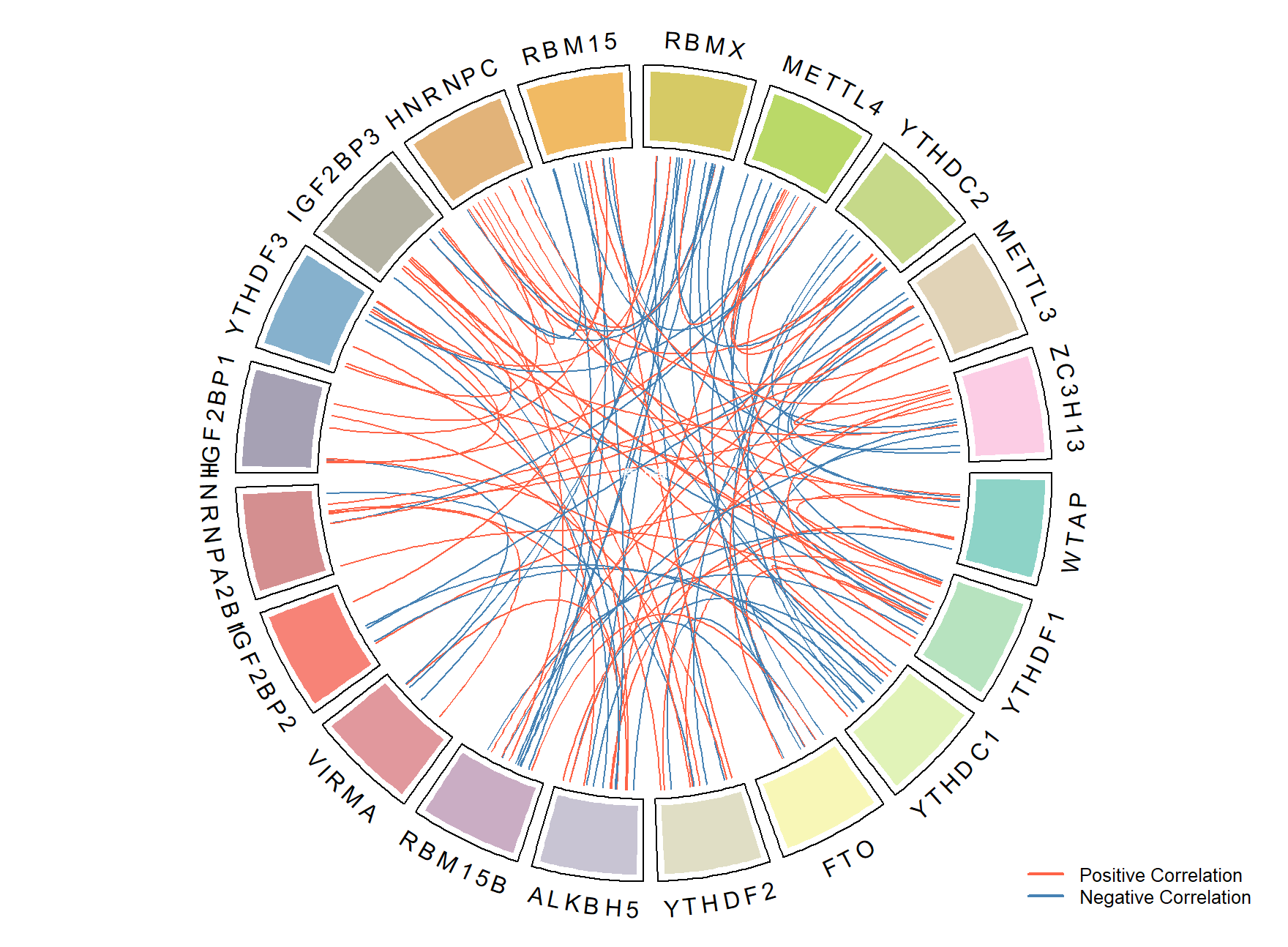
这是一幅基因相关性圈图,通常被称为 Circos 图,它用于可视化多个基因之间的相关性关系。包括以下结构:
外围环:每个外部的长方形块代表一个基因,每个基因都有其对应的颜色来区分。
连线:圈内的线条代表基因之间的相关性:
红色连线表示基因之间存在正相关性。
蓝色连线表示基因之间存在负相关性。
线条密集程度:线条密集区域意味着存在大量相关性强的基因对。
图例:右下角图例指出了颜色所代表的相关性方向——红色为正相关,蓝色为负相关。
2.数据要求
基因列表:需要一组基因名称,通常在图中外围部分进行标识。
基因间相关性:每对基因之间的 Spearman 相关性系数,需要指定其值以判断其是正相关还是负相关。
显著性水平:需要确定哪些基因对之间的相关性达到统计显著性,才能在图中展示连线。
颜色标注:定义正负相关性的颜色,通常正相关为红色,负相关为蓝色。
3.用R代码生成虚拟数据
# 安装和加载必要的包
if (!require('circlize')) install.packages('circlize')
if (!require('dplyr')) install.packages('dplyr')
if (!require('ComplexHeatmap')) install.packages('ComplexHeatmap')
if (!require('RColorBrewer')) install.packages('RColorBrewer')
library(circlize)
library(dplyr)
library(ComplexHeatmap)
library(RColorBrewer)
# 生成虚拟的基因相关性数据
set.seed(123) # 设置随机种子以确保可重复性
genes <- c('WTAP', 'YTHDF1', 'YTHDC1', 'FTO', 'YTHDF2', 'ALKBH5', 'RBM15B', 'VIRMA', 'IGF2BP2', 'HNRNPA2B1', 'IGF2BP1', 'YTHDF3', 'IGF2BP3', 'HNRNPC', 'RBM15', 'RBMX', 'METTL4', 'YTHDC2', 'METTL3', 'ZC3H13')
# 创建一个包含基因相关性的虚拟数据框
gene_pairs <- expand.grid(genes, genes) %>% filter(Var1 != Var2)
correlation_values <- runif(nrow(gene_pairs), min = -1, max = 1)
correlation_data <- data.frame(
gene1 = gene_pairs$Var1,
gene2 = gene_pairs$Var2,
correlation = correlation_values
)
# 将相关性数据保存为CSV文件
write.csv(correlation_data, file = 'gene_correlation_data.csv', row.names = FALSE)
gene_correlation_data <- correlation_data
4.绘图
# 读取CSV文件
gene_correlation_data <- read.csv('gene_correlation_data.csv')
gene_correlation_data <- correlation_data
# 筛选出显著相关的基因对(假设绝对值大于0.7的相关性为显著)
significant_data <- gene_correlation_data %>% filter(abs(correlation) > 0.7)
# 定义颜色映射
link_colors <- ifelse(significant_data$correlation > 0, "#FF6347", "#4682B4")
sector_colors <- colorRampPalette(brewer.pal(8, "Set3"))(length(genes))
# 绘制基因相关性圈图
circos.clear() # 清除之前的circos设置
circos.par(gap.degree = 2) # 设置每个扇区之间的间隙
circos.initialize(factors = genes, xlim = c(0, 1))
# 绘制每个基因的扇区并添加颜色
circos.trackPlotRegion(
factors = genes, ylim = c(0, 1), panel.fun = function(x, y) {
circos.text(CELL_META$xcenter, CELL_META$ylim[2] + mm_y(5), CELL_META$sector.index, facing = "bending.inside", niceFacing = TRUE)
circos.rect(CELL_META$xlim[1], 0, CELL_META$xlim[2], 1, col = sector_colors[CELL_META$sector.numeric.index], border = NA)
}
)
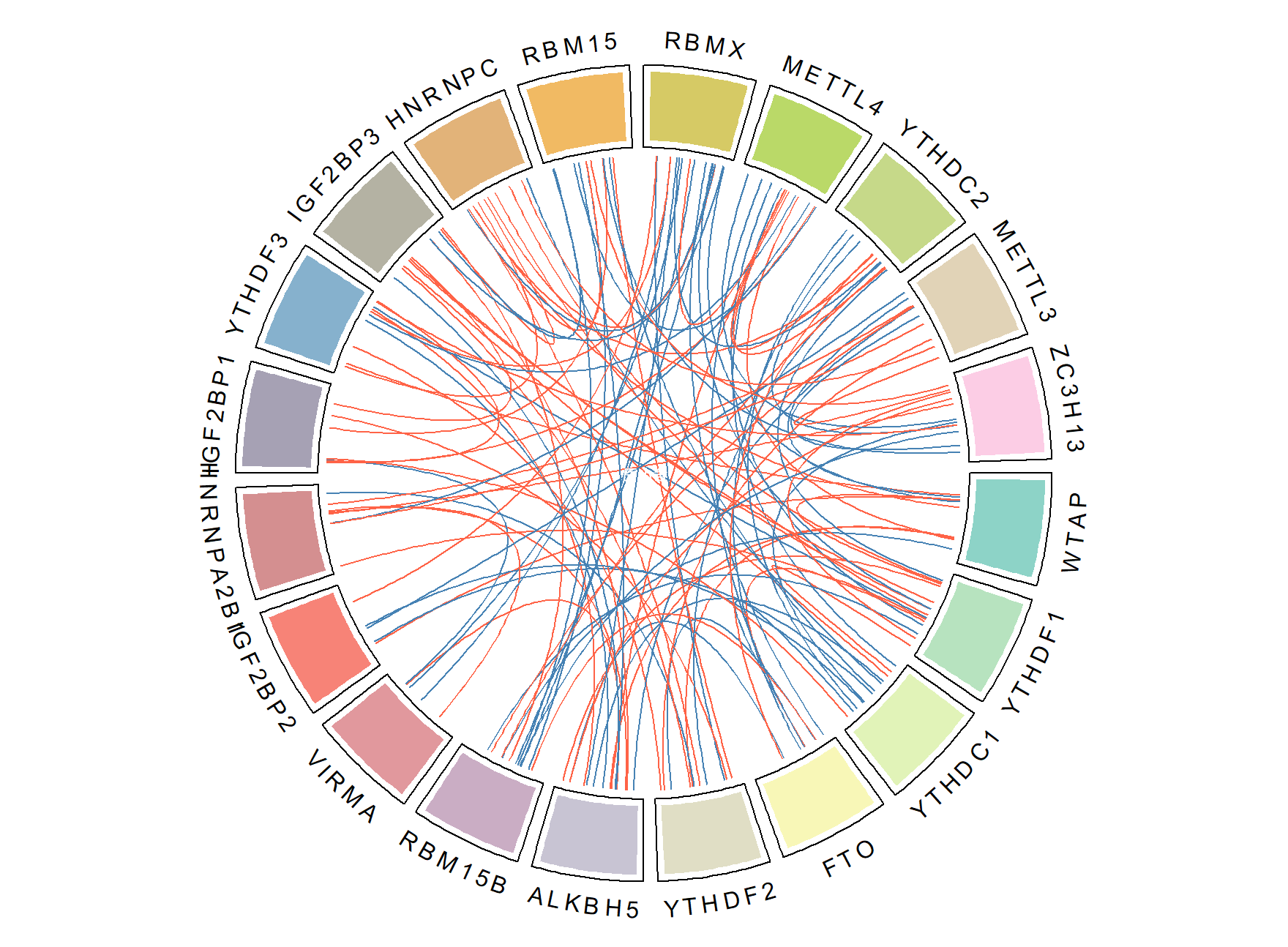
# 绘制基因之间的相关性连线
for (i in 1:nrow(significant_data)) {
circos.link(
significant_data$gene1[i], runif(1),
significant_data$gene2[i], runif(1),
col = link_colors[i], border = NA
)
}
# 添加图例
legend("bottomright", legend = c("Positive Correlation", "Negative Correlation"),
col = c("#FF6347", "#4682B4"), lty = 1, lwd = 2, bty = "n", cex = 0.8)

阅读剩余
版权声明:
作者:Q
链接:https://yunshangtulv.com.cn/?p=813
文章版权归作者所有,未经允许请勿转载。
THE END